NGSure低丰度变异检测定制化服务
基于qPCR和Sanger测序的高性能低丰度变异检测定制化设计服务
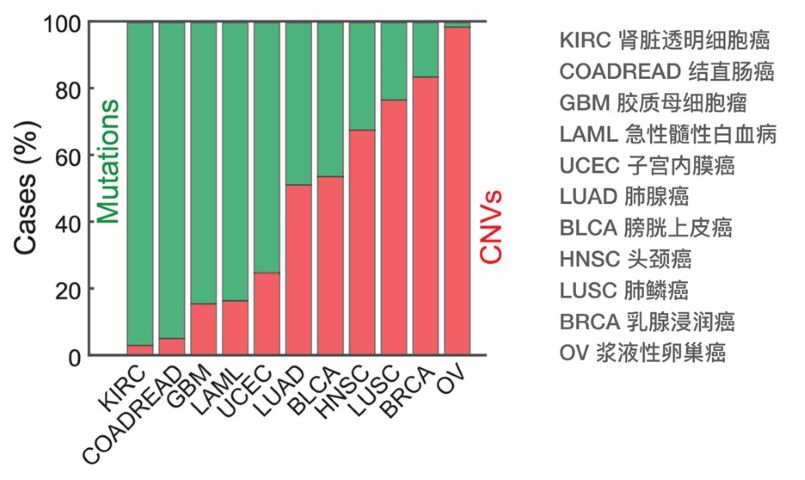
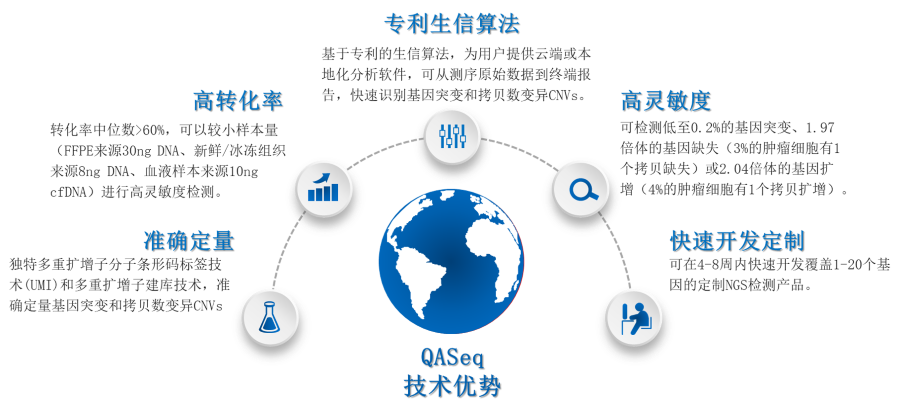
QASeq(Quantitative Amplicon Sequencing)是一种全新的定量扩增子测序技术,可以帮助研发人员实现NGS检测产品的快速开发定制化(4-8周),基因的缺失和扩增,也称为拷贝数变异(CNVs)存在于多种肿瘤中,基因突变与拷贝数变异可以作为肿瘤预后标志物和治疗靶点,具有重要临床意义。QASeq可在FFPE样本、新鲜或冰冻组织样本或血液cfDNA样本中,检测到突变丰度低至0.2%的基因突变和1.97倍数的缺失或2.04倍数的基因扩增。
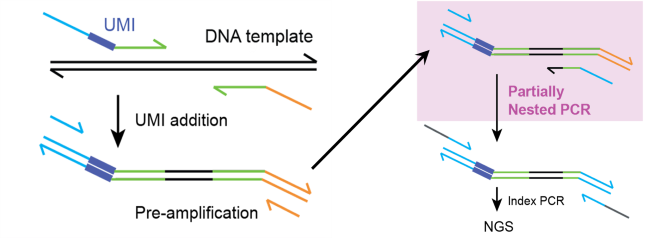

QASeq利用独特的多重扩增子分子条形码标签技术(UMI)和多重扩增子建库技术,可实现准确定量FFPE组织、新鲜或冰冻组织或血液cfDNA样本中的基因突变和拷贝数变异检测,该技术有较高的转化率和灵敏度,搭配专利生信算法,可实现定制NGS产品的快速开发。(专利号:CN202080013877.8)

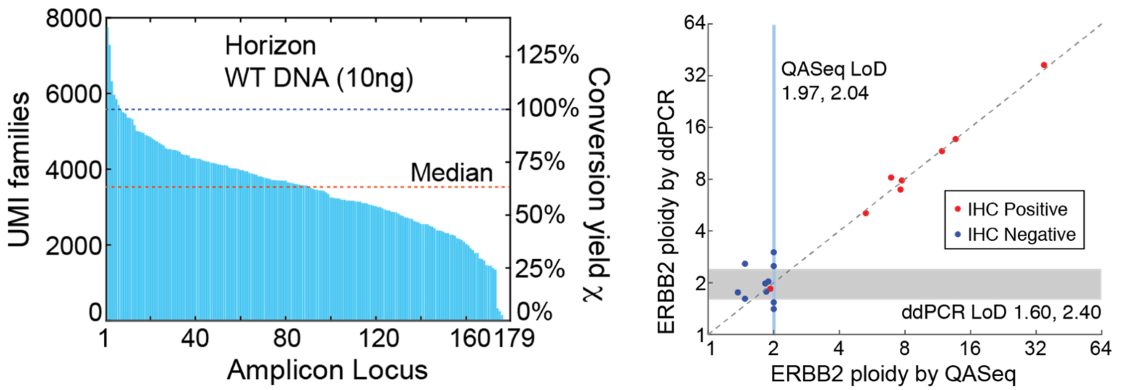
QASeq技术通过使用不同的UMI标记DNA分子的两条链,因此具有较高的转化率,中位数>60%(以10 ng Horizon Multiplex I野生型cfDNA参考标准)。在肿瘤CNV检测中的检出限要高于FISH等传统方法,如Foundation One CNV检出限为 2.6 ploidy,而QASeq技术通过优化捕获/扩增区间,结合UMI标签,肿瘤CNV检测的检出限最低能达到2.05(HER2)

目前,QASeq已广泛应用于样本起始量低,需用进行PCR扩增的项目如单细胞,cfDNA,ctDNA等。在肿瘤液体活检中,癌症患者ctDNA变异检测面临两个问题,一是起始量低,往往只有几十ng(相比传统DNA文库500ng~1μg起始),二是ctDNA在cfDNA中的比例很低,(波动范围较大,0.01%~90%,受肿瘤分期、转移等影响,大部分在千分之几的数量级),导致cfDNA中能检测到的肿瘤突变频率很低,有研究表明半数以上突变频率在0.4%以下。目前常用的ctDNA检测技术主要由ARMS-qPCR,ddPCR,NGS等方法。其中NGS由于检测范围广,能同时检测突变、扩增、融合,其应用范围更广。然而为了提高检测灵敏性,在NGS检测中需要对原始样本进行PCR扩增富集(PCR扩增错误引入,duplicate增加),同时提高测序深度(液体活检测序深度往往大于10,000X,扩增错误放大),因此会产生大量背景噪音,导致低频突变难以识别,限制了测序的准确度。通过QASeq技术可有效减少扩增带来的背景噪音,从而提高检测的特异性。